Ou seja, pessoas que nunca tiveram contato com o SARS-CoV-2, mas ao longo da vida foram expostas a outros vírus, como os que causam resfriado comum, apresentam alguma resposta imune a ele. Porque as células de defesa têm memória.
Os cientistas suspeitam que isso pode explicar – em parte – por que a covid-19 se desenvolve com mais ou menos gravidade em diferentes pessoas, faixas etárias etc. (+)
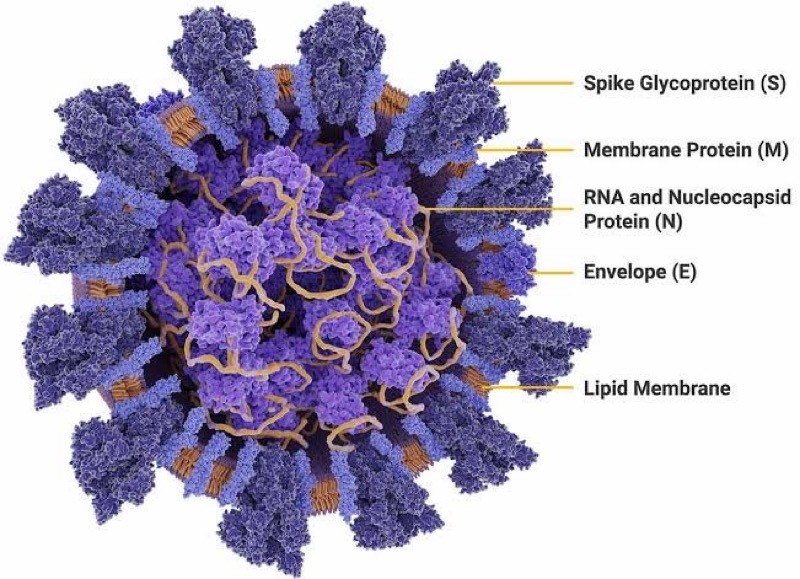
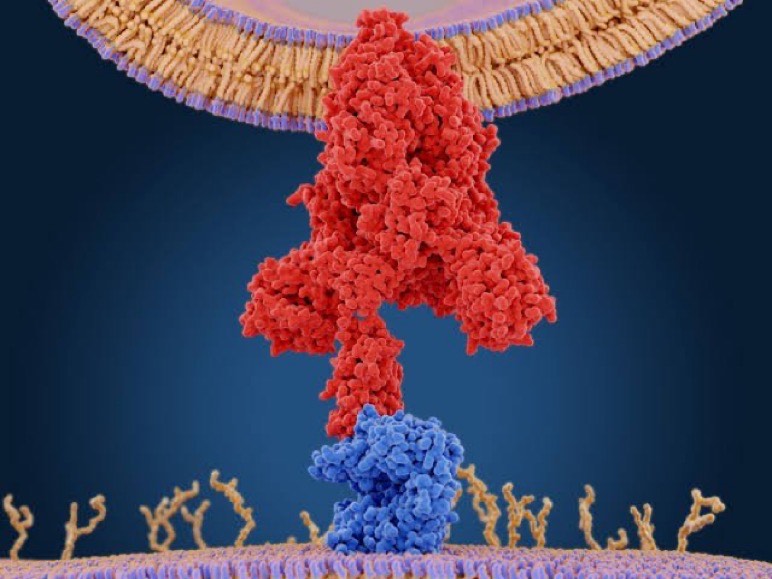
No experimento, feito com amostras de sangue, as respostas mais fortes das células T reativas ao coronavírus foram associadas à proteína spike do vírus, aquela que parece um espinho, e é usada por ele para invadir as células humanas. (+)
Por Luiza Caires
Epítopos de células T SARS-CoV-2 reativas, seletivas e reativas
Resumo
Existem muitas incógnitas sobre as respostas imunes humanas ao vírus SARS-CoV-2. Células T CD4 + reativas à SARS-CoV-2 foram relatadas em indivíduos não expostos, sugerindo memória pré-existente de células T reativas cruzadas em 20-50% das pessoas. No entanto, a fonte dessas células T tem sido especulativa. Usando amostras de sangue humano derivadas antes da descoberta do vírus SARS-CoV-2 em 2019, mapeamos epítopos de células T 142 no genoma SARS-CoV-2 para facilitar o interrogatório preciso do repertório de células T CD4 +específico para SARS-CoV-2 . Demonstramos uma gama de memória CD4 + pré-existenteCélulas T que são reativas cruzadas com afinidade comparável à SARS-CoV-2 e aos coronavírus comuns do resfriado HCoV-OC43, HCoV-229E, HCoV-NL63 ou HCoV-HKU1. Assim, a memória de células T variegadas para os coronavírus que causam o resfriado comum pode estar subjacente a pelo menos parte da extensa heterogeneidade observada na doença de COVID-19.
O surgimento da SARS-CoV-2 no final de 2019 e sua subsequente disseminação global levou a milhões de infecções e morbimortalidade substancial ( 1 ). A doença de coronavírus 2019 (COVID-19), a doença clínica causada pela infecção por SARS-CoV-2, pode variar de doença autolimitada leve a síndrome do desconforto respiratório agudo e morte ( 2 ). Os mecanismos subjacentes ao espectro dos estados de gravidade da doença de COVID-19 e a natureza da imunidade protetora contra o COVID-19 atualmente não são claros.
Estudos que dissecam a resposta imune humana contra SARS-CoV-2 começaram a caracterizar respostas de células T específicas ao antígeno SARS-CoV-2 ( 3 – 8 ), e vários estudos descreveram ativação acentuada de subconjuntos de células T em pacientes agudos com COVID-19 ( 9 – 13 ). Surpreendentemente, estudos de células T específicos para antígenos, realizados com cinco coortes diferentes, relataram que 20-50% das pessoas que não haviam sido expostas à SARS-CoV-2 apresentaram reatividade significativa das células T direcionadas contra peptídeos correspondentes às seqüências de SARS-CoV-2 ( 3). – 7) Os estudos foram de coortes geograficamente diversas (EUA, Holanda, Alemanha, Cingapura e Reino Unido), e o padrão geral observado foi que a reatividade das células T encontrada em indivíduos não expostos era predominantemente mediada por células T CD4 + . Especulou-se que esse fenômeno possa ser devido a respostas de memória preexistentes contra coronavírus (HCoVs) “resfriado comum” humano, como HCoV-OC43, HCoV-HKU1, HCoV-NL63 ou HCoV-229E. Esses HCoVs compartilham homologia de sequência parcial com SARS-CoV-2, circulam amplamente na população em geral e são geralmente responsáveis por sintomas respiratórios leves ( 14 – 16) No entanto, a hipótese de imunidade reativa cruzada entre SARS-CoV-2 e HCoVs para resfriado comum ainda aguarda ensaios experimentais. Essa potencial imunidade de células T reativas cruzadas preexistente ao SARS-CoV-2 tem amplas implicações, pois poderia explicar aspectos de resultados clínicos diferenciais do COVID-19, influenciar modelos epidemiológicos de imunidade do rebanho ( 17 , 18) ou afetar o desempenho do COVID -19 vacinas candidatas.
Repertório do epítopo em indivíduos não expostos à SARS-CoV-2
Para definir o repertório de células T CD4 + que reconhecem epítopos de SARS-CoV-2 em indivíduos não expostos anteriormente, usamos estimulação in vitro de PBMCs por 2 semanas com conjuntos de peptídeos de 15 mer. Este método é conhecido por ser robusto para detectar respostas de células T de baixa frequência a alérgenos, antígenos bacterianos ou virais ( 19 , 20 ), incluindo células T ingênuas ( 21 ). Para a triagem de epítopos SARS-CoV-2, utilizamos amostras de PBMC de indivíduos não expostos coletados entre março de 2015 e março de 2018, muito antes da ocorrência da circulação global de SARS-CoV-2. Os sujeitos não expostos foram confirmados soronegativos para SARS-CoV-2 (fig. S1A).
As células T reativas à SARS-CoV-2 foram expandidas com um conjunto de peptídeos que abrangem toda a sequência da proteína spike (CD4-S), ou um “megapool” (CD4-R) não spike (CD4-R) de epítopos previstos do non spike regiões (isto é, “restante”) do genoma viral ( 4) No total, foram rastreados 474 peptídeos de 15-mer SARS-CoV-2. Após 14 dias de estimulação, a reatividade das células T contra “mesopools” intermediários, cada uma compreendendo aproximadamente 10 peptídeos, foi testada usando um ensaio FluoroSPOT (por exemplo, 22 mesopools CD4-R; fig. S2A). Os mesopolos positivos foram ainda desconvoluídos para identificar epítopos individuais específicos de SARS-CoV-2. Os resultados representativos de um doador mostram a deconvolução dos mesopools P6 e P18 para identificar sete epítopos diferentes de SARS-CoV-2 (fig. S2B). Os ensaios intracelulares de coloração de citocinas (ICS) específicos para IFN-γ determinaram se as células T específicas do antígeno que respondiam aos mesopools SARS-CoV-2 eram CD4 + ou CD8 +Células T (fig. S2C). Os resultados das 44 combinações de doador / mesopool CD4-R e 40 doador / mesopool CD4-S que produzem uma resposta positiva são mostrados na fig. S2, D e E, respectivamente. Em 82/88 casos (93,2%), as células que responderam à estimulação da mesopool SARS-CoV-2 eram claramente células T CD4 + , conforme julgado pela razão de células que respondem a CD4 / CD8. Em quatro casos (4,5%), as células que responderam foram células T CD8 + e em dois casos (2,3%) as respostas foram mediadas por células T CD4 + e CD8 + . O fato de que as células T CD8 + raramente foram detectadas não foi surpreendente, uma vez que os peptídeos usados no CD4-R englobavam epítopos previstos da classe II e o CD4-S é constituído por peptídeos de 15 meros (peptídeos de 9 a 10 meros são ideais para CD8+ Células T). Além disso, o protocolo de reestimulação de duas semanas foi originalmente projetado para expandir as células T CD4 + ( 20 ). No geral, estes resultados indicaram que a estratégia de triagem de peptídeos utilizava epítopos de SARS-CoV-2 mapeados reconhecidos por células T CD4 + em indivíduos não expostos.
Um total de 142 epítopos de SARS-CoV-2 foram identificados, 66 da proteína spike (CD4-S) e 76 do restante do genoma (CD4-R) (tabela S1). Para cada combinação de epítopo e doador respondente, inferiram-se possíveis restrições de HLA com base na capacidade prevista de ligação ao HLA do epítopo específico para os alelos específicos de HLA presentes no doador respondente ( 22 ). Cada doador reconheceu uma média de 11,4 epítopos (intervalo de 1 a 33, mediana 6,5; fig. S3A). Quarenta dos 142 epítopos foram reconhecidos por 2 ou mais doadores (fig. S3B), representando 55% da resposta total (fig. S3C). Esses 142 epítopos de SARS-CoV-2 mapeados podem ser úteis em estudos futuros como reagentes para rastrear células T CD4 + em indivíduos infectados com SARS-CoV-2 e em ensaios de vacina com COVID-19.
Distribuição do epítopo por ORF de origem
Embora uma ampla variedade de antígenos SARS-CoV-2 diferentes tenha sido reconhecida, foi impressionante que vários dos epítopos que produzem as respostas mais frequentes (isto é, reconhecidas em vários doadores) ou mais vigorosos (isto é, a maioria das células SFC / 10 6 ) foram derivado do antígeno de pico SARS-CoV-2 (tabela S1). Portanto, avaliamos a distribuição geral dos epítopos das células T 142 mapeados entre todas as proteínas SARS-CoV-2, em comparação com o tamanho relativo de cada antígeno SARS-CoV-2 ( Fig. 1, A e B ). Notavelmente, 54% da resposta positiva total foi associada a epítopos derivados de spike ( Fig. 1A ; 11% para RBD e 44% para a porção não-RBD da spike). De relevância para o desenvolvimento da vacina COVID-19, apenas 20 %das respostas de pico foram derivadas da região do domínio de ligação ao receptor (RBD) ( Fig. 1A ; comparando 11% vs 44%, como descrito acima), e a região RBD representou apenas 11 % da reatividade geral das células T CD4 + ( Fig. 1A ). Os epítopos mapeados foram distribuídos de maneira bastante uniforme pelo genoma de SARS-CoV-2 na proporção do tamanho de cada proteína ( Fig. 1B ; p = 0,038, r = 0,42). Além das fortes respostas direcionadas ao pico, também foram observadas respostas para ORF6, ORF3a, N, ORF8 e dentro do Orf1a / b, onde nsp3, nsp12, nsp4, nsp6, nsp2 e nsp14 foram reconhecidos com maior destaque. Esses resultados epítopos mapeados no nível ORFeome se sobrepõem parcialmente aos ORFs segmentados por CD4 +Células T nos casos COVID-19 ( 4 ). Notavelmente, nenhum epítopo derivado da proteína da membrana (M) foi identificado em indivíduos não expostos ( Fig. 1B ), mas M é reconhecido de maneira robusta pelas respostas das células T CD4 + específicas para SARS-CoV-2 nos casos de COVID-19 ( 4 ). A falta de epítopos de classe II de qualidade em M não era surpreendente, com base na biologia molecular de M; M é uma pequena proteína com três domínios transmembranares. Combinados, os dados indicam que os epítopos da classe II estão relativamente disponíveis no genoma SARS-CoV-2, mas que as células T CD4 + com memória SARS-CoV-2 têm como alvo preferencial proteínas altamente expressas durante a infecção, exemplificadas por M e S (pico) epítopo mapeando resultados.

Homologia de sequência dos epítopos SARS-CoV-2 identificados com outros HCoVs comuns
Quando este estudo de mapeamento de epítopos foi iniciado, uma suposição era de que o mapeamento de epítopos de cultura de células T in vitro revelaria um repertório de epítopos associado à geração de novo de respostas de células T ingênuas. No entanto, enquanto esses estudos de mapeamento de epítopos estavam em andamento, nós e outros detectamos reatividade ex vivo significativa contra pools em massa de peptídeos SARS-CoV-2 ( 3 – 7 ). Especulamos que isso possa refletir a presença de células T de memória reativas cruzadas entre os coronavírus humanos comuns (HCoVs) e o SARS-CoV-2. Esses outros HCoVs circulam amplamente nas populações humanas e são geralmente responsáveis por doenças respiratórias leves, geralmente não diagnosticadas, como o resfriado comum ( 14 – 16).) No entanto, atualmente há uma falta de dados experimentais que abordem se as células T CD4 + de memória , reativas cruzadas entre SARS-CoV-2 e outros HCoVs, realmente existem.
Portanto, determinamos a seguir o grau de homologia para todos os quatro HCoVs de circulação ampla para todos os 142 epítopos de SARS-CoV-2 aqui identificados. Para a análise, dividimos os peptídeos em três grupos com base na imunogenicidade: 1) nunca imunogênicos, 2) imunogênicos em um indivíduo ou 3) imunogênicos em dois ou mais indivíduos ( Fig. 1C) Houve similaridade de sequência significativamente maior nos peptídeos reconhecidos por mais de um indivíduo em comparação com os peptídeos reconhecidos por um único indivíduo ou nenhum (p <0,0001, teste de Mann Whitney bicaudal). Além disso, quase todos os doadores da coorte não exposta utilizados para o rastreio do epítopo eram soropositivos para três HCoVs de coronavírus comum em circulação (HCoV-NL63, HCoV-OC42, HCoV-HKU1) (fig. S1B). Assim, os dados de homologia epitópica e soropositividade sugerem que a reatividade cruzada de células T era plausível entre SARS-CoV-2 e HCoVs já estabelecidos na população humana.
Para selecionar subconjuntos de epítopos a serem analisados em mais detalhes, plotamos a magnitude da resposta das células T de cada epítopo positivo por doador ( Fig. 1D ). Esta análise confirma a dominância do antígeno de pico sobre os epítopos derivados do restante do genoma (p <0,001, teste de Mann Whitney bicaudal).
Em seguida, selecionamos duas categorias de epítopos de interesse SARS-CoV-2. A primeira categoria foi epitopos com potencial reatividade cruzada de HCoVs. Inicialmente, selecionamos o corte arbitrário de 67%, uma vez que concluímos que um 9mer é a região epitópica envolvida na ligação à classe II ( 23 ), e que geralmente um ou dois resíduos além da região central de 9 méres são necessários para otimizar reconhecimento ( 24 ) ( Fig. 1D , vermelho). Segundo, filtramos independentemente quaisquer epítopos associados a altas respostas (top ~ 30%; Fig. 1Dazul). Isso resultou na seleção de 31 epítopos do pico (6 com alta homologia e 25 para respostas dominantes), organizados em um novo conjunto de CD4- [S31]. Da mesma forma, geramos um novo pool de CD4- [R30], composto por 30 epítopos do restante do genoma (9 com alta homologia e 21 associados a fortes respostas; Fig. 1D ). Estes conjuntos de epítopos foram então utilizados para estudos adicionais de células T CD4 + .
Evidência direta de reatividade aos epitopos do HCoV homólogos aos epitopos do SARS-CoV-2
Para abordar diretamente se a reatividade contra SARS-CoV-2 em doadores não expostos pode ser atribuída à reatividade cruzada contra outros HCoVs, projetamos um pool de peptídeos que engloba peptídeos homólogos aos epítopos CD4-R30, derivados de HCoV-229E, HCoV-NL63, HCoV -OC43, HCoV-HKU1 e vários outros HCoVs (consulte Métodos), para um total de 129 homólogos de HCoV (HCoV-R129; tabela S2). Da mesma forma, sintetizamos um pool que englobava peptídeos homólogos ao pool de epítopos SARS-CoV-2 CD4-S31, consistindo em epítopos potenciais derivados de outros HCoVs, para um total de 124 homólogos de HCoV (HCoV-S124; tabela S3).
Em seguida, utilizamos um teste de marcador induzido por ativação (ensaio AIM, ( 25 – 27 )) para detectar células T específicas de vírus em um novo conjunto de doadores não expostos, não utilizados para os estudos de identificação de epítopos ( Fig. 2A e tabela S4), e um conjunto de pacientes convalescentes com COVID-19 (tabela S5). Detectamos respostas significativas de células T CD4 + ex vivo contra os peptídeos SARS-CoV-2 não spike (CD4-R) e spike (CD4-S) em comparação ao controle negativo (DMSO) ( Fig. 2, B e C ; p <0,0001 ep <0,0001, respectivamente, Mann-Whitney bicaudal). Essas respostas foram aumentadas nos casos COVID-19 em comparação com indivíduos não expostos ( Fig. 2D ; p = 0,0015 ep = 0,0022, respectivamente, Mann-Whitney bicaudal), conforme relatado anteriormente (4 ) Nos indivíduos não expostos,foram detectadasfrequências significativas de célulasTCD4 + contra os conjuntos de epítopos CD4-R30 e CD4-S31 SARS-CoV-2 em comparação com o controle negativo ( Fig. 2B ; p = 0,0063 ep = 0,0012, respectivamente, Mann-Whitney bicaudal). Tambémfoi observada reatividadesignificativa de célulasTCD4 + contra os conjuntos HCoV-R129 e HCoV-S124 correspondentes de peptídeos homólogos correspondentes de outros HCoVs ( Fig. 2D ; p <0,0001 ep <0,0001, Mann-Whitney bicaudal). A detecção de célulasTCD4 + com pools de peptídeos selecionados com base na homologia foi consistente com a hipótese de que célulasTCD4 + reativas cruzadasentre SARS-CoV-2 e outros HCoVs existem em muitos indivíduos.

A reatividade contra CD4-R30 e CD4-S31 ( Fig. 2D ; p = 0,0008 ep = 0,0026, respectivamente), mas não contra HCoV-R129 e HCoV-S124, foi aumentada em casos de COVID-19 em comparação com indivíduos não expostos ( Fig. 2C ). Assim, a reatividade pré-existente de células T CD4 + a epítopos de HCoV é modulada por COVID-19 e a exposição a epítopos de reatividade cruzada SARS-CoV-2 em COVID-19. Esses dados dos casos de COVID-19 não sustentam a hipótese de que a exposição ao HCoV possa induzir um fenômeno original do pecado antigênico, prejudicando as respostas subsequentes das células T aos epítopos SARS-CoV-2 ( 28 , 29 ), pelo menos nos casos de COVID-19 de gravidade média da doença.
Em seguida, examinamos o fenótipo de memória ex vivo das células T, respondendo aos vários megapools do epítopo. Os resultados de um doador não exposto representativo são mostrados na Fig. 3A . As células respondentes em doadores não expostos foram predominantemente encontradas na população de células T CD4 + com memória efetiva (CD45RA neg CCR7 neg ), seguidas pelas células T de memória central (CD45RA neg CCR7 pos ) ( 30 ) ( Fig. 3, A, B e D ) Padrões comparáveis de células efetoras e de memória central foram observados entre as células T CD4 + específicas do antígeno detectadas nos casos de COVID-19 ( Fig. 3, C e D ). Em conclusão, o CD4+ Células T em dadores não expostos que reconhecem SARS-CoV-2 epitopos, e epitopos de outros HCoVs, tem um fenótipo de memória. No geral, esses dados são consistentes com as células T CD4 + reativas a SARS-CoV-2 em indivíduos não expostos, sendo células T CD4 + de memória específica de HCoV com reatividade cruzada a SARS-CoV-2.

Identificação de epítopos SARS-CoV-2 reativos cruzados com outros HCoVs comuns
Os epítopos derivados dos conjuntos CD4-R30 e CD4-S31 foram utilizados para gerar linhas de células T de curto prazo derivadas por estimulação de PBMCs de indivíduos não expostos. As PBMCs foram estimuladas com um epítopo cognato individual de SARS-CoV-2 demonstrado ser reconhecido pelas células T desse sujeito ( Fig. 1 e tabela S1). No geral, as linhas de células T podem ser derivadas específicas para um total de 42 epítopos de SARS-CoV-2.
Essas linhas de células T foram testadas em seguida quanto à reatividade cruzada contra vários homólogos de coronavírus, análoga a uma abordagem previamente bem-sucedida em estudos de flavivírus ( 31 ). A reatividade cruzada entre o reconhecimento do epítopo SARS-CoV-2 e outro reconhecimento do epítopo HCoV foi detectada em 10/42 (24%) das linhas de células T ( Fig. 4, A a J ). A reatividade cruzada foi associada a epítopos derivados do pico de SARS-CoV-2, N, nsp8, nsp12 e nsp13. Em três casos, os análogos do HCoV eram antígenos melhores que o peptídeo SARS-CoV-2, sugerindo que eles podem ser o imunogênio cognato ( Fig. 4, E, I e J) Um epítopo de pico de SARS-CoV-2 foi testado em dois doadores diferentes, com resultados semelhantes, sugerindo que os padrões de reatividade cruzada de HCoV são recorrentes entre indivíduos. Também são mostradas linhas de células T SARS-CoV-2 com reatividade cruzada ( Fig. 4, K a L e fig. S4). É possível que a reatividade cruzada com esses epítopos possa ser detectada se as linhas de células T de indivíduos adicionais forem testadas. Além disso, esses epítopos podem ser homólogos de alguma outra sequência viral ainda não identificada ou reconhecida por células T ingênuas cognatas que se expandem na cultura in vitro ( 32 ). Além disso, apenas 3/18 casos de epítopos de resposta fortes (definidos na Fig. 1D) foram reativos, em comparação com 4/5 dos epítopos mais fracos (p = 0,02, teste exato de Fisher). Para demonstrar ainda mais que as respostas reativas cruzadas em doadores não expostos são de fato derivadas de células T de memória, estimulamos a memória purificada e células T CD4 + ingênuas com o pool de epítopos CD4- [S31]. Após 14 dias, detectamos respostas ao pool de peptídeos CD4- [S31] a partir de culturas de células T CD4 + com memória , mas não de células T CD4 + ingênuas (fig. S8). Em suma, esses dados demonstram que as células T CD4 + de memória que reconhecem coronavírus comuns ao resfriado, incluindo HCoV-OC43, HCoV-HKU1, HCoV-NL63 e HCoV-229E, podem exibir reatividade cruzada substancial ao epítopo homólogo em SARS-CoV-2.

Em seguida, examinamos, para cada par de epítopos SARS-CoV2: HCoV, o grau de homologia da sequência de aminoácidos e qualquer relação entre homologia e reatividade cruzada de células T, considerando diferentes faixas de homologia potencialmente relevante. Apenas 1% (1/99) de pares de peptídeos com 33-40% de homologia foram reativos. Na faixa de homologia de 47-60% do epítopo, observamos reatividade cruzada em 21% dos casos (7/33). Surpreendentemente, a homologia do epítopo ≥ 67% foi associada à reatividade cruzada em 57% dos casos (21/37; p = 0,0001 ou p = 0,0033 pelo teste exato de Fisher bicaudal, quando comparado com os epítopos da faixa de 33-40% ou 47 -60%, respectivamente). Foi observada uma relação entre a homologia do epítopo e a reatividade cruzada das células T CD4 + . Os dados demonstraram que a seleção arbitrária utilizada conforme descrito na Fig. 1D, foi de fato suportado pelos dados experimentais. Assim, ~ 67% de homologia de aminoácidos parece ser uma referência útil para a consideração de potencial reatividade cruzada entre epítopos de classe II. Em resumo, aqui identificamos mais de 140 epítopos de células T humanas derivados de todo o genoma da SARS-CoV-2. Fornecemos evidências diretas de que numerosas células T CD4 + que reagem aos epítopos SARS-CoV-2 realmente reagem de maneira cruzada com as sequências homólogas correspondentes de qualquer um dos vários HCoVs diferentes em circulação comum e que essas células reativas são amplamente células T CD4 + de memória canônica . Essas descobertas sobre as especificidades das células T de HCoV reativas cruzadas contrastam fortemente com os anticorpos neutralizantes de HCoV, que são específicos da espécie para HCoV e não mostraram reatividade cruzada contra a SARS-CoV-2 RBD (33 – 35 ). Com base nesses dados, é plausível supor quea memória das células THCoV CD4 + reativa cruzada preexistenteem alguns doadores possa ser um fator contribuinte para variações nos resultados da doença de pacientes com COVID-19, mas atualmente é altamente especulativo ( 36). )
FONTE: Revista Science
 SAÚDE NA CAPITAL SUA FONTE DE INFORMAÇÃO SEGURA SOBRE SAÚDE
SAÚDE NA CAPITAL SUA FONTE DE INFORMAÇÃO SEGURA SOBRE SAÚDE